
Bioinformatica
I servizi bioinformatici Bio-Fab sono pensati per rispondere alla crescente richiesta da parte dei nostri clienti di un servizio completo di analisi, dalla generazione del dato alla elaborazione e trasformazione. Ogni specifica analisi è pensata per rispondere alle esigenze del cliente che sarà partecipe, assieme ai nostri esperti, dell'elaborazione del dato scientifico prodotto nei laboratori Bio-Fab. I servizi bioinformatici Bio-Fab sono pensati per offrire una consulenza sia pre che post analisi, sia sui dati ottenuti dai nostri sequenziantori che su dati consegnati direttamente dal cliente. Le analisi, in ciascuno dei servizi e con la massima flessibilità, possono essere di tipo base, avanzata e/o personalizzate.
Alcuni servizi bioinformatici Bio-Fab riguardano:
- Analisi Metagenomica (Metabarcoding)
- Assemblaggio di genomi interi (Whole genome seq; plasmid finder; virulence finder..)
- Espressione differenziale di trascritti (mRNA sequencing)
- Espressione diferenziale dei miRNA

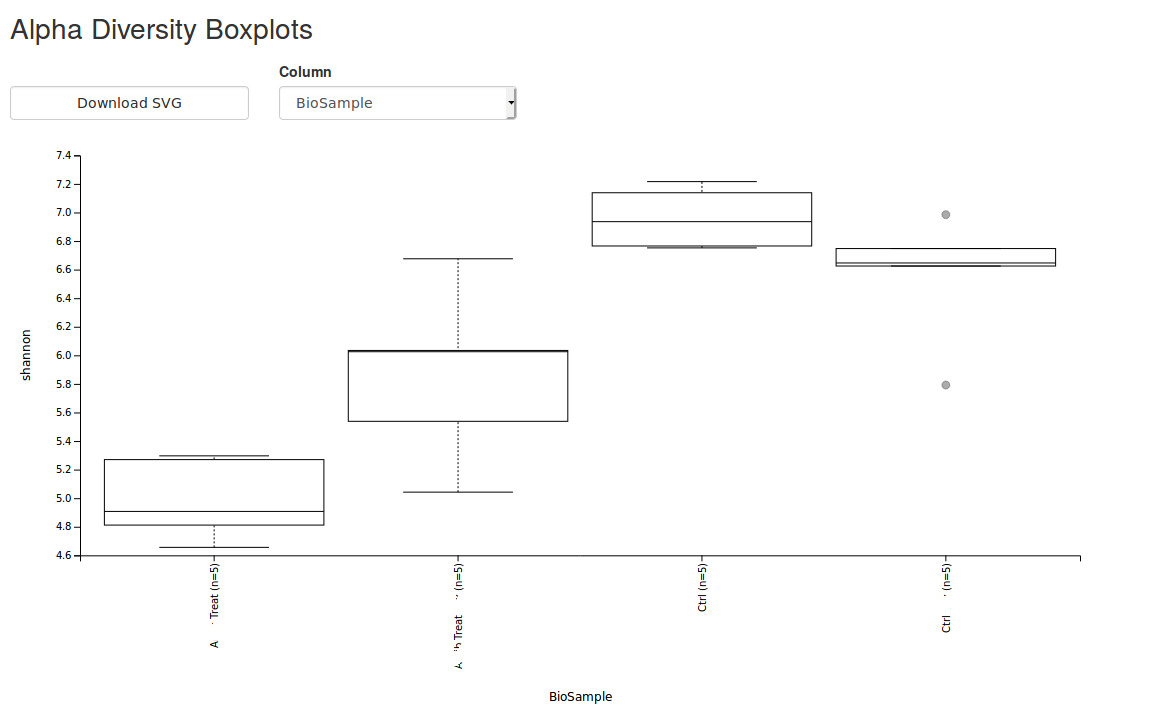

Bio-Fab Services
Word Clouds

COVERAGE: Che cosa significa nel contesto NGS?
Il termine “Coverage” è usato spesso, ma a volte è causa di confusione, perché oltre alla definizione comune ci sono descrizioni alternative, vari tipi (comprese varie forme di calcolo) e confusione con un altro termine: la profondità del sequenziamento (sequencing depth).
Il termine "Coverage" in NGS descrive sempre una relazione tra reads (frammenti sequenziati) e un riferimento (Reference, ad esempio un intero genoma, un locus o una posizione), a differenza della profondità di sequenziamento che descrive il numero di reads totali. È importante distinguere tra i loro significati:
- Coverage in termini di ridondanza: numero di reads che si allineano o "coprono" un riferimento. Descrive con quale frequenza, in media, ciascuna base di una sequenza di riferimento è coperta dalle reads allineate. Questa è un'informazione importante perché sono necessarie più osservazioni di una base in una data posizione per ottenere una chiamata affidabile (ad esempio nelle analisi di varianti). Pertanto il coverage in termini di ridondanza viene anche utilizzato come unità per la significatività statistica dei dati di sequenziamento. A seconda del riferimento (intero genoma o un locus), ci sono diversi modi per calcolare il coverage.
- Coverage in termini di percentuale di copertura di un riferimento da parte delle reads ad esso allineate. Per esempio, se il 90% di un riferimento è coperto dalle reads (e il 10% no) diciamo che abbiamo una copertura del 90%.
- Profondità di sequenziamento: numero totale di frammenti sequenziati (raw reads) o prodotti dalla piattaforma di sequenziamento (di solito espressa in milioni di reads).
Tra questi termini, l’unico che è possibile stimare a priori è la profondità di sequenziamento (sequencing depth). La stima delle altre due forme di Coverage è dipendente dall’allineamento delle reads al riferimento oltre che da parametri di qualità usati per il filtraggio delle raw reads durante i primi passaggi dell’analisi bioinformatica. Tutti e tre i significati attribuibili al termine Coverage sono stimati attraverso la stessa formula, riarrangiabile per ciascuna terminologia.
Qual'è un buon coverage per un progetto NGS?
Esistono delle linea guida generali o specifiche che descrivono il Coverage ottimale per un progetto di sequenziamento. Dipende fortemente dal tipo di esperimento, dalla specie, dal materiale di input, dalla piattaforma di sequenziamento e da altri fattori. Tuttavia, ti consigliamo di contattare il nostro servizio di sequenziamento per questa domanda prima di ordinare un progetto, in modo da poter sfruttare l'esperienza esistente.
Ci sono regioni nel genoma che non sono coperte dal sequenziamento del DNA?
Se il genoma fosse una strada e tu creassi una mappa delle orme dei passanti su quella strada, la mappa avrebbe certamente delle aree bianche. In altre parole: alcune regioni genomiche non possono essere coperte molto bene sequenziando il DNA con la tecnologia NGS a causa di sequenze ripetute (ad esempio ripetizioni in tandem) che sono abbondanti in un'ampia gamma di specie.
Circa il 50% del genoma umano è composto da ripetizioni. Le ripetizioni sono una sfida per i programmi di allineamento e assemblaggio delle sequenze soprattutto se vengono generate reads corte, molto simili. Può essere paragonato a un grande puzzle in base al quale alcuni pezzi del puzzle si adattano in diversi punti. Quindi le ripetizioni creano ambiguità nell'allineamento e nell'assemblaggio, che, a loro volta, possono produrre distorsioni ed errori nell'interpretazione dei risultati.
Anche la %GC influenza il coverage in generale. Le quattro basi (ACTG) normalmente non sono equamente distribuite in un genoma. Le regioni di DNA con %GC alta e bassa sono difficili da amplificare a causa di una stabilità più elevata rispetto a una regione di DNA con un contenuto di basi misto. In questi casi, la DNA polimerasi è incline a produrre artefatti. Questi effetti disturbano le fasi di amplificazione richieste nella maggior parte dei protocolli. Di conseguenza, i frammenti di regioni con %GC alto / basso sono sottorappresentati, con un coverage scarso e sbilanciato. Alcuni progetti attuali cercano di migliorare il sequenziamento di tali regioni minimizzando gli artefatti prodotti dalla PCR (amplificazione nel caso di piattaforme come Illumina) o rinunciando alla fase di amplificazione (esempio con MinION).
Infine la frammentazione, nei metodi shotgun, influenza il Coverage, perché come riportato in letteratura, la frammentazione del DNA è un processo in gran parte non casuale, specialmente per i metodi di taglio meccanico. Ciò porta a un Coverage non uniforme di varie regioni genomiche e può risultare in regioni con zero o basso coverage rispetto a quanto stimato/desiderato.
I Nostri Servizi
Bioinformatica
© 2006-2024 BIO-FAB RESEARCH - ALL RIGHTS RESERVED
Registered office: Bio-Fab Research srl – Via Mario Beltrami, 5 – 00135 ROMA
P.I./C.F./CCIAA n. 08736731004 - REA n. 1114841 - Cap. soc. i.v. 10.000,00

