
FAQs
Come vengono sintetizzati gli oligo?
Gli oligonucleotidi vengono sintetizzati usando un sintetizzatore di DNA programmabile costituito fondamentalmente da un sistema di reagenti per la sintesi di oligonucleotidi.
La prima base viene fissata ad un supporto solido, solitamente una biglia di vetro o polistirene che ancora la catena di DNA in formazione nella colonna di reazione.
La sintesi del DNA consiste in una serie di reazioni chimiche:
- I fase - Deblocking: la prima base, fissata al supporto solido per mezzo di un linker molecolare, viene de-protetta per rimozione di un gruppo Trityl protettivo. Questa fase genera un gruppo 5’OH che reagisce con la base successiva.
- II fase - Coupling: la base successiva è attivata e si accoppia al gruppo 5’OH dell’ultima base della catena.
- III fase - Capping: ogni base che non reagisce viene protetta da un “cap” per evitare che non prenda parte alle reazione delle fasi successive del ciclo di sintesi.
- IV fase - Oxidation: il legame tra la prima base e la seconda base accoppiata con successo viene ossidato per stabilizzare la catena in formazione.
- V fase - Deblocking: il gruppo 5’Trityl viene rimosso dalla base alla quale è stato aggiunto.
- Ciascun ciclo di reazione da come risultato l’aggiunta di una singola base di DNA. Una catena di basi di DNA può essere costruita grazie alla ripetizione di diversi cicli di sintesi fino ad ottenere la lunghezza desiderata.
Cos’è la scala di sintesi?
La dicitura “scala di sintesi” fa riferimento alla quantità iniziale di monomero CPG (controller-pore glass) legato al supporto che viene usato per iniziare la sintesi del DNA, e non alla quantità di materiale finale sintetizzato. Quando viene specificata una scala di 40 nmole, significa che vengono inserite approssimativamente 40 nmoles della prima base nel sintetizzatore. Per la sintesi di un 25-mer, almeno il 25% del materiale di partenza finirà in sequenze fallite. Ciò significa che non è possibile produrre 40 nmoles di prodotto full-length da una scala di sintesi di 40 nmole. Le perdite avvengono durante le fasi di sintesi, processamento post-sintetico, trasferimento di materiale e controllo di qualità.
Per oligonucleotidi con lunghezza standard (<= 33 nucleotidi) viene garantita una certa resa di DNA (OD260 applicata a 20 mer) che viene distribuita in un range di +/- 20% relativamente alla rispettiva scala di sintesi e metodo di purificazione.
Cos’è l’efficienza di accoppiamento (coupling efficiency)?
L’efficienza di accoppiamento viene usata per misurare l’efficienza del sintetizzatore nell’aggiungere nuove basi alla catena di DNA in formazione. Se ogni base disponibile sulla catena ha reagito con successo con la nuova base in ingresso, l’efficienza di accoppiamento sarà il 100%. Soltanto alcune reazione raggiungono il 100% di efficienza. Durante la sintesi del DNA, il valore massimo di efficienza di accoppiamento che si può ottenere è normalmente attorno al 99%. Questo significa che ad ogni step di accoppiamento approssimativamente l’1% delle basi disponibili non riescono a reagire con la nuova base in ingresso. L’efficienza di accoppiamento è significativamente influenzata dalla qualità delle materie prime utilizzate (amiditi e solutioni), dalle condizioni dello strumento, e dal protocollo di sintesi utilizzato.
Perché è importante l’efficienza di accoppiamento?
L’efficienza di accoppiamento è importante perchè gli effetti sono cumulativi durante la sintesi del DNA. La tabella 1 mostra gli effetti dell’efficienza di accoppiamento sulla percentuale di prodotti full-length al termine della sintesi di oligonucleotidi di lunghezza differente.

Anche con un oligonucleotide relativamente corto di 20 basi, l’1% di differenza nell’efficienza di accoppiamento può significare il 15% medio in più di DNA presente in seguito alla sintesi di prodotti full-length.
La tabella mostra inoltre che più lungo è un oligonucleotide, minore sarà la resa di un prodotto full-length attesa. Questo fenomeno è dovuto al tipo di chimica utilizzata. Anche se si ottiene il 99% di efficienza di accoppiamento per ogni aggiunta di una singola base, il prodotto grezzo di sintesi di un 95 mer sarà costituito soltanto dal 38,5% di oligonucleotide full-length.
La separazione dei prodotti full-length per HPLC comporta una perdita addizionale e quindi rese più basse.
Cosa è il gruppo tritile?
Ogni base aggiunta durante la sintesi possiede un gruppo protettore dimetossi-tritil attaccato alla posizione 5’-ossidrile. Questo gruppo acido protegge la base da reazioni chimiche indesiderate durante il ciclo di sintesi e viene rimosso al primo step di ciascun ciclo di sintesi immediatamente prima dell’aggiunta di una nuova base, fino alla fine della fase di allungamento. Il gruppo tritile finale viene mantenuto oppure viene rimosso, a seconda del metodo di purificazione scelto.
Come si misura l’efficienza di accoppiamento?
Il gruppo tritile è senza colore quando è attaccato ad una base di DNA ma restituisce una caratteristica colorazione arancio quando rimosso. L’intensità di questo colore può essere misurata per mezzo di spettrofotometria UV ed è direttamente correlata al numero di molecole di tritile presenti. In seguito al primo step di accoppiamento, la quantità di tritile rilasciato durante il deblocking è direttamente proporzionale alla quantità di oligonucleotidi full-length sintetizzato nel ciclo precedente. Dalla comparazione delle intensità del tritile prodotto dopo il primo ed ultimo accoppiamento, si può calcolare il successo medio di accoppiamento di basi per ciclo e quindi l’efficienza di accoppiamento.
Quando faccio un grande ordine di oligo spesso la consegna ritarda, perché?
La sintesi del DNA è un processo complicato che ha ottenuto un significativo miglioramento negli ultimi 10 anni. Nonostante questo miglioramento, tutti i produttori hanno un tasso di fallimento consistente. Noi stiamo sviluppando nuovi processi e sistemi per minimizzare questi fallimenti. E’comunque inevitabile che occasionalmente ci troviamo costretti a ri-sintetizzare alcuni oligonucleotidi.
E’necessario purificare gli oligo?
Dipende se sono state richieste modifiche o meno e da quale sarà l’applicazione. Sequenze fallimentari possono essere generate sia durante il processo di sintesi che durante il processo di post-sintesi. Noi raccomandiamo di richiedere che tutte le modifiche siano purificate per HPLC. Per la purezza raccomandata e la scala, vedi la tabella sotto.
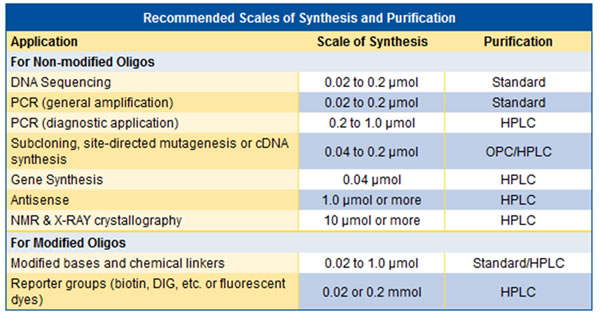
Perché gli oligo richiedono purificazioni?
Finita la sintesi del DNA, la catena completa viene rilasciata dal supporto solido in seguito a trattamento con una soluzione basica come idrossido di ammonio. Dopo il trattamento si ottiene una nuova soluzione Questa contenente l’oligonucleotide full-lenght richiesto più tutte le catene di DNA che sono abortite durante la sintesi. Se un 20mer è stato sintetizzato, la soluzione dovrebbe contenere anche i 19mer abortiti, i 18mer abortiti, i 17mer abortiti ecc., in piccole quantità.
La quantità di sequenze abortite presenti è influenzata dall’efficienza di accoppiamento e dalla lunghezza dell’oligo (Tabella 1). Queste sequenze abortite possono competere con il prodotto full-length in alcune applicazioni come sublconing, gene synthesis, antisense. Ricorda che in generale, più elevato il grado di purezza, minore sarà la resa finale!
Perché ci sono rese più basse per oligo modificate?
Molte degli amiditi modificati sono meno stabili e non accoppiano così efficientemente come le basi non modificate, dunque le sequenze abortite sono più abbondanti che in una sintesi normale. Di conseguenza, la maggior parte degli oligonucleotidi modificati dovrebbe essere purificata per HPLC per rimuovere la quantità maggiore di sequenza abortite. Come risultato si hanno delle rese più basse sebbene il prodotto finale, è molto più puro e la probabilità che il prodotto ottenuto sia totalmente modificato è maggiore.
I miei oligo hanno un gruppo fosfato al 5 primo?
A meno di richieste specifiche, gli oligonucleotidi vengono sintetizzate senza 3’ né 5’ fosfato.
I miei oligo annilano ma non ligano. Perché?
Le reazioni di ligazioni richiedono un 5’ fosfato. Se i tuoi oligo non contengono un 5’ fosfato, la ligazione non avverrà o avverrà con un’efficienza molto bassa. Il problema può essere risolto senza ordinare un altro oligonucleotidi: fosforila i tuoi oligonucleotidi enzimaticamente con una chinasi prima di usarli per la reazioni di ligazione.
Perché non c’è una resa per la scala di sintesi di 1 um cinque volte più alta della scala di sintesi da 0,2 um?
Per la scala da 0,2, il monomero di accoppiamento è fatto ad un eccesso di moli 40-50 volte. Farlo per scale di sintesi più alte, sarebbe proibitivo a livello di costi. Le sintesi su larga scala sono fatte solo ad un eccesso di moli 10 volte superiore di amiditi e le colonne 1 umol non sono state completamente ottimizzate ad oggi in termini di flusso, spazio morto e diffusione. Comunque, per aumentare le rese di scale di sintesi maggiori, i tempi di accoppiamento vengono estesi ad aumentare le efficienze di accoppiamento.
Qual è la lunghezza massima di sintesi per un oligo?
Tutto dipende dal potere risolutivo del metodo di purificazione e dall’efficienza di accoppiamento del sintetizzatore. Noi possiamo sintetizzare oligo di 180 basi e ottenere quantità accettabili per la purificazione in HPLC. In ogni caso, maggiore è la lunghezza dell’oligonucleotide, maggiore saranno le possibilità di accumulare errori di sequenza.
Anche la composizione in basi e le scale di sintesi sono possono essere fattori determinanti. La tabella 1 mostra che con un’efficienza di accoppiamento al 99%, una soluzione cruda di 95-mers dovrebbe contenere il 38% di prodotto full-length and il 62% di sequenze abortite. E’ necessario prendere in considerazione anche gli effetti dei trattamenti chimici, come la depuri nazione. La depurinazione mostra principalmente effetti sulla base A. La frequenza di depuri nazione è bassa ma potrebbe aumentare significativamente con l’allungarsi del primer.
Per queste ragioni, indichiamo 80 basi di lunghezza massima, che riteniamo essere la lunghezza che può essere sintetizzata economicamente senza grandi problemi. Poiché la probabilità che la sintesi di oligo più lunghi debba essere ripetuta a causa di una interruzione di sintesi prematura dovuta a basse efficienze di accoppiamento, i prezzi per oligonucleotidi maggiori di 80 nucleotidi sono più elevati.
In cosa rispospendo i miei oligonucleotidi, e a quale concentrazione?
In acqua purificata, TE o qualunque altro buffer biologico accettato come diluente. Il volume raccomandato di diluente è tra 100 ul e 1 ml, la concentrazione è dipendente dall’applicazione per cui devono essere usati gli oligo e la resa del prodotto finale. La concentrazione standard per PCR primers è di 0,1 mM.
Quanto è stabile il mio oligonucleotidi dopo che l’ho risospeso?
Se viene usato un diluente sterile, sarà stabile a 20°C per diversi giorni o settimane, a 4°C per circa un mese. Se conservato a -20°C o -70°C, sarà stabile per diversi mesi, addirittura anni. Il ripetersi di congelamento-scongelamento deve essere evitato, altrimenti l’oligonucleotide sarà denaturato.
Per oligonucleotidi modificati – specialmente per quelli marcati con sonde fluorescenti – in aggiunta a quanto detto sopra, sarebbe necessario ridurre al minimo l’esposizione alla luce a causa dell’effetto bleaching. Noi raccomandiamo anche di conservare gli oligonucleotidi marcati con sonde a concentrazioni elevate e di non diluirli se non è previsto l’uso entro 24 ore. Maggiore è il fattore di diluizione, più veloce avverrà la decadenza della fluorescenza. Inoltre, è possibile aliquotarli, sciogliendo un’aliquota per volta, diluendo l’aliquota appena prima dell’uso e conservando la diluizione al buio a 4°C. Se un oligonucleotide è liofilizzato e non viene risospeso, può essere conservato chiuso a temperatura ambiente oppure per un tempo indeterminato a -20°C. Se l’oligonucleotide viene risospeso, sarà necessario conservarlo a -20°C o a temperature più basse.
Ci sono delle linee guida da seguire nel disegno degli oligonucletidi?
Sì e sono le seguenti:
Lunghezza della sequenza – solitamente vengono sintetizzati oligonucleotidi dalle 5 alle 180 o più basi. La maggior parte delle sequenze vanno dalle 18 alle 30 basi con una media di 24 basi. Bisogna ricordare che più lungo è l’oligo, minore è la percentuale di prodotto full-length nella sintesi cruda comportando la riduzione delle rese dopo la purificazione.
Composizione della sequenza – è necessario assicurarsi che la sequenza non dia luogo a strutture a forcina. Più di 6 basi ripetute consecutivamente (ad esempio GGGGGGG) possono essere problematiche poiché riducono le rese finali.
Modificazioni – Quando possibile, è necessario scegliere modificazioni al 5’ terminale. La sintesi del DNA avviene automaticamente in direzione 3’-> 5’. Ogni aggiunta di nucleotide è efficiente al 98-99% con una percentuale dell’1-2% di oligo troncato e cappato ad ogni posizione. Posizionando la modificazione al 5’ terminale si è sicuri che soltanto l’oligonucleotide full length risulta modificato. Inoltre, poiché la maggior parte delle modificazioni sono più idrofobiche dell’oligonucleotide non modificato, l’oligo full-length modificato si lega più strettamente alla fase inversa durante la purificazione per HPLC.
Scala di sintesi – Il termine “scala di sintesi” si riferisce alla quantità di supporto solido derivatizzato usato. La quantità finale di prodotto ricavato dipenderà dalla lunghezza della sequenza, dalla sequenza della struttura secondaria, dal tipo di modificazione scelta, dal posizionamento della modificazione, dal numero delle modificazioni per nucleotide e dal metodo di purificazione usato.
Metodo di purificazione – è necessario scegliere il metodo di purificazione sulla base del livello di purezza richiesto dalla specifica applicazione.
Perché il MALDI-TOF?
Nella procedura di produzione degli oligo http, ogni singolo oligonucleotide viene normalmente analizzato per MALDI TOF per ottenere lo spettro di massa e compararlo allo spettro di massa teorico atteso. La documentazione della procedura di controllo di qualità http può essere fornita su richiesta con un costo aggiuntivo.
I Nostri Servizi
Oligonucleotidi
© 2006-2024 BIO-FAB RESEARCH - ALL RIGHTS RESERVED
Registered office: Bio-Fab Research srl – Via Mario Beltrami, 5 – 00135 ROMA
P.I./C.F./CCIAA n. 08736731004 - REA n. 1114841 - Cap. soc. i.v. 10.000,00

